【Stringtieの使い方】RNA-Seq解析における遺伝子発現量の定量
はじめに
次世代シーケンサーを用いてRNA-Seq解析を行うと、FASTQファイルと呼ばれる生データが得られます。生データを参照配列にマッピングした後に、それぞれの遺伝子にマッピングされたリードをカウントすることによって遺伝子発現量を定量していきます。
本ページではマッピング結果から新規アイソフォームの同定とアイソフォームごとの発現量を推定するソフトウェアであるStringTieの使い方を説明します。
インストール
こちらにコンパイル済みバイナリが用意されていますので、これをダウンロードしていきます。(Macでバージョン2.2.1を使用する場合)
ヘルプを表示してみます。
以下のように表示されればインストール成功です。
新規アイソフォームの同定
以下のコマンドで新規アイソフォームの同定とアイソフォームごとの発現量の推定を行います。まずは、sample1, sample2, sample3, sample4 の4つのサンプルについてそれぞれ実施します。
-Gオプションを使用してリファレンスを指定することで、アイソフォームの同定の際のガイドとしています。
sample1.gtf ~ sample4.gtf としてそれぞれのサンプルの結果が得られました。
ファイルの結果ファイルの中身は以下のようになっています。
| カラム | 説明 |
| 1カラム目 | 染色体名 |
| 2カラム目 | 必ず「StringTie」が入る |
| 3カラム目 | feature type(exon, transcript, mRNA, 5'UTR等) |
| 4カラム目 | featureの開始位置(1-based index) |
| 5カラム目 | featureの終了位置(1-based index) |
| 6カラム目 | 必ず1000が入る |
| 7カラム目 | 転写産物の向き |
| 8カラム目 | 必ず「.」が入る |
| 9カラム目 | セミコロン区切りの追加情報 |
追加情報には以下の情報がセミコロン「;」区切りで入っています。
マージ
サンプルごとに結果が得られましたが、アイソフォームがサンプルごとに異なっており、比較ができなくなっています。そのため以下のコマンドでこれらの結果をマージしていきます。
マージされたアノテーションファイル「merged.gtf」が出力されました。
アイソフォームごとの発現量の推定
最後にマージされたアノテーションファイルをもとにアイソフォームごとの発現量の推定を行なっていきます。
-eオプションを使用することで、新規アイソフォームの同定は行わずに、merged.gtfに含まれるアイソフォームのみを解析対象としています。 また、-BオプションでBallgown用のファイル(*.ctab)も出力するようにしています。Ballgownは発現変動遺伝子を抽出するためのソフトウェアです。
DESeq2やedgeR用のファイルの準備
上記の手順でBallgown用のファイルは得られましたが、このファイルではDESeq2やedgeRでは解析を行うことができません。
すべてのサンプルの結果をまとめたCSVファイルを作成して、DESeq2やedgeRでも解析を行えるようにしていきます。
この操作には、prepDE.py3を用います。prepDE.py3はprepDE.pyがPython3に対応したものですのでprepDE.pyを使用しても同様です。
今回はリード長が150 bpですので -l 150 のようにオプションを指定しています。
これにより以下のような「gene_count_matrix.csv」と「transcript_count_matrix.csv」というファイルが得られました。
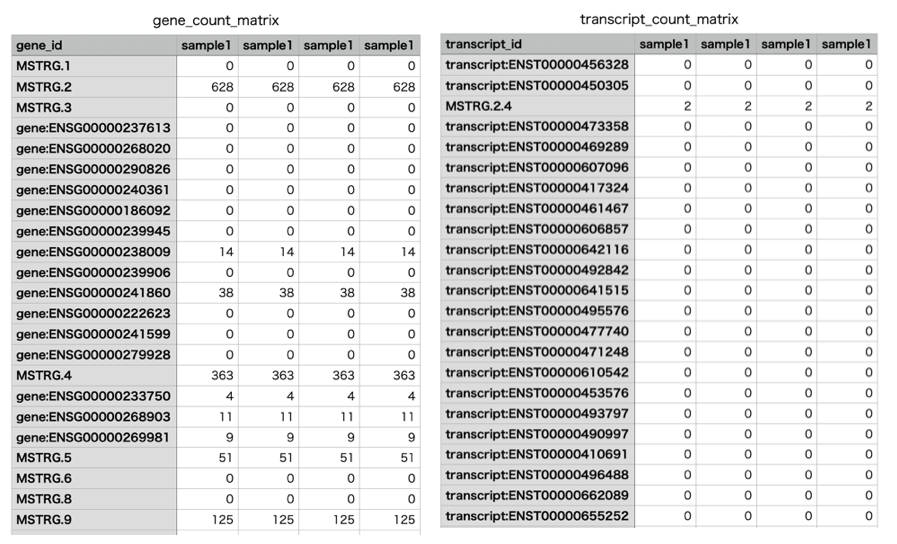
https://github.com/gpertea/stringtie/issues/126で議論されていますが、ペアリードの場合にリードカウントを合計しても実際のリードの本数とは一致しないようですが、このまま使用するしかなさそうです。
論文に必要な解析が簡単にできるRNA-Seqデータ解析ツール
RNA-Seqデータ解析ツールを利用すれば、外部委託や共同研究者への依頼は必要ありません。高スペックなコンピュータの準備やLinuxコマンドの操作も不要ですので、いますぐにご自身で解析できるようになります。
遺伝子発現量の定量、発現変動遺伝子抽出(DEG解析)、Volcano plot描画、MAプロット描画、ヒートマップ描画、GO解析、パスウェイ解析等 を簡単に実施できます。