【TPM】RNA-Seq解析における遺伝子発現量の補正方法
はじめに
RNA-Seq解析によって得られたリードのカウント結果は、そのままでは遺伝子間やサンプル間での比較を行うことができません。
なぜなら、遺伝子の長さが長いほどその遺伝子にマッピングされるリードは多くなりますし、シーケンスにより得られた総リード数が多ければ多いほどそのサンプルのそれぞれの遺伝子にマッピングされるリードが多くなるためです。
そのため様々な補正方法が提案されていますが、本ページではTPMについて説明します。以前は、FPKM/RPKMという指標も多く用いられていましたが、 適切に遺伝子発現量を表現できていないと指摘があり、現在はTPMが用いられることが多くなってきています。
TPMの定義
TPMは「transcripts per million」の略で、FPKM/RPKMの代替として提案された補正方法です。
マッピングされた全リード数を100万、転写産物長を1000塩基になるように補正する点ではFPKM/RPKMと同様ですが、補正する順番が異なり、先に長さの補正をしてから総リード数による補正を行います。
計算式は以下の通りです。(\(q_i\) はマッピングされたリード数、\(l_i\) は転写産物長を表します。)
FPKMを用いて、以下のように表現することもできます。
Effective length
TPMの計算方法はソフトウェアによって少しずつ異なっています。 例えば、ソフトウェアによっては\(l_i\)に実際の転写産物長を使用するのではなく、Effective lengthを使用することがあります。
Effetive Lengthは以下で計算できます。
\(μ_{FLD}\)は平均フラグメント長を示しています。
Effective lengthをTPMの計算に使用することで、より適切に長さの影響を補正することができると言われています。
論文に必要な解析が簡単にできるRNA-Seqデータ解析ツール
RNA-Seqデータ解析ツールを利用すれば、外部委託や共同研究者への依頼は必要ありません。高スペックなコンピュータの準備やLinuxコマンドの操作も不要ですので、いますぐにご自身で解析できるようになります。
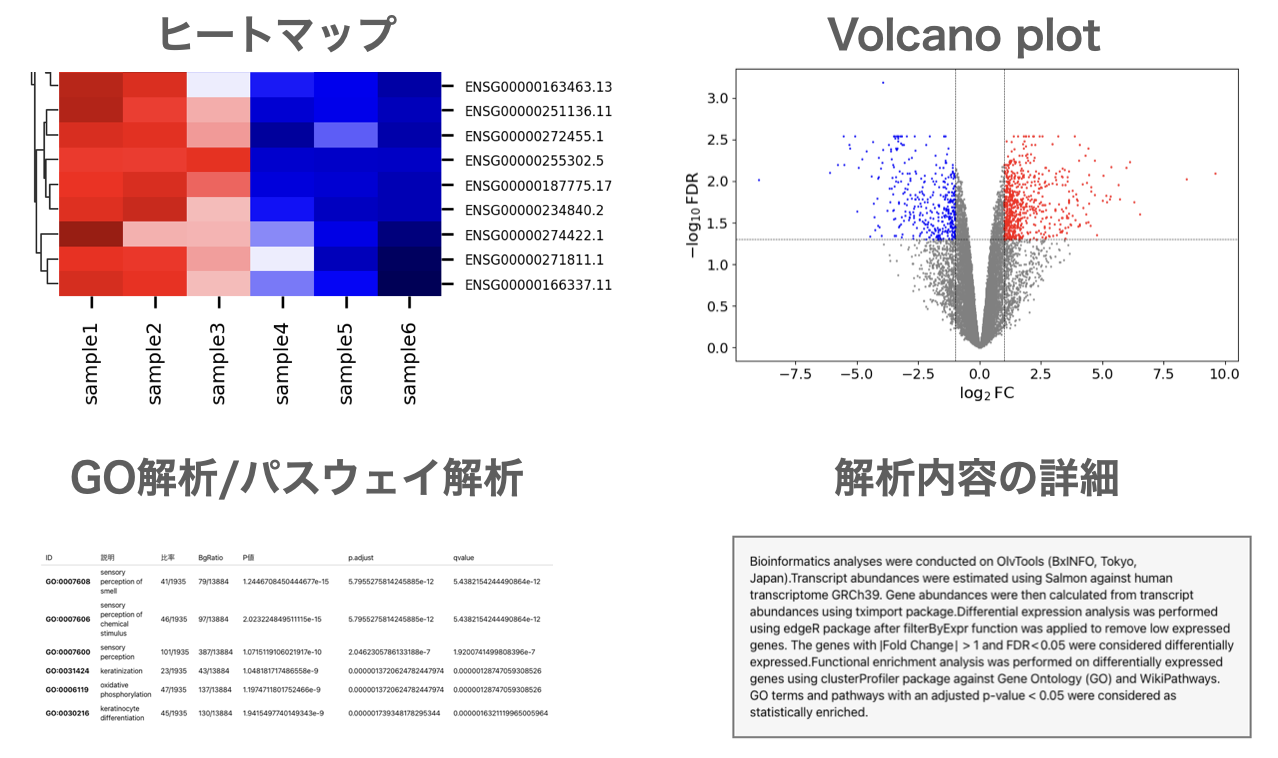
遺伝子発現量の定量、発現変動遺伝子抽出(DEG解析)、Volcano plot描画、MAプロット描画、ヒートマップ描画、GO解析、パスウェイ解析等 を簡単に実施できます。