>
【Bowtie2の使い方】RNA-Seq解析におけるマッピング
【Bowtie2の使い方】RNA-Seq解析におけるマッピング
更新日: 2026/1/20
はじめに
RNA-Seq解析によって得られたシーケンスデータを用いて遺伝子発現量を定量していく際に、一般的にマッピング処理が行われます。 マッピングとはリード配列 (FASTQファイル)を参照配列の一致する箇所に並べていく処理のことを言います。 RNA-Seqにおけるマッピングによく使われるソフトウェアとして、HISAT2、STAR、 Bowtie2といったものがあります。 本ページではBowtie2の使い方を説明します。
RNA-Seqのデータ解析の一連の流れはこちらをご覧ください。
インストール
condaを利用してインストール可能です。
$ conda install -c bioconda bowtie2
ヘルプを表示してみます。
$ bowtie2 -h
以下のような内容が表示されれば成功です。
bowtie2 -h
Bowtie 2 version 2.4.1 by Ben Langmead (langmea@cs.jhu.edu, www.cs.jhu.edu/~langmea)
Usage:
bowtie2 [options]* -x <bt2-idx> {-1 <m1> -2 <m2> | -U <r> | --interleaved <i> | -b <bam>} [-S <sam>]
<bt2-idx> Index filename prefix (minus trailing .X.bt2).
NOTE: Bowtie 1 and Bowtie 2 indexes are not compatible.
<m1> Files with #1 mates, paired with files in <m2>.
Could be gzip'ed (extension: .gz) or bzip2'ed (extension: .bz2).
<m2> Files with #2 mates, paired with files in <m1>.
Could be gzip'ed (extension: .gz) or bzip2'ed (extension: .bz2).
<r> Files with unpaired reads.
Could be gzip'ed (extension: .gz) or bzip2'ed (extension: .bz2).
<i> Files with interleaved paired-end FASTQ/FASTA reads
Could be gzip'ed (extension: .gz) or bzip2'ed (extension: .bz2).
<bam> Files are unaligned BAM sorted by read name.
<sam> File for SAM output (default: stdout)
<m1>, <m2>, <r> can be comma-separated lists (no whitespace) and can be
specified many times. E.g. '-U file1.fq,file2.fq -U file3.fq'.
...
Index作成(build)
まず以下のコマンドで参照配列のindexを作成していきます。
$ bowtie2-build -f genome.fa genome
genome.faはマッピングしたい参照配列のFASTAファイルです。gzipで圧縮されたままでも問題ありません。
この操作によりgenome.1.bt2〜genome.4.bt2とgenome.rev.1.bt2、genome.rev.2.bt2の6つのファイルが作成されます。 indexファイルは文字列を高速に検索するために必要なファイルで、Bowtie2に限らずほぼすべてのマッピングソフトウェアにおいて事前作成が必要です。
マッピング
次に、リード配列を参照配列にマッピングしていきます。
$ bowtie2 -x genome -1 reads1.fastq.gz -2 reads2.fastq.gz -S output.sam
この操作によりSAMファイルが出力されました。SAMファイルはマッピング結果のファイルですが、BAMファイルに変換してソートしておくと便利なので以下の操作を行います。
$ samtools view -bS output.sam > output.bam
$ samtools sort output.bam > output.sorted.bam
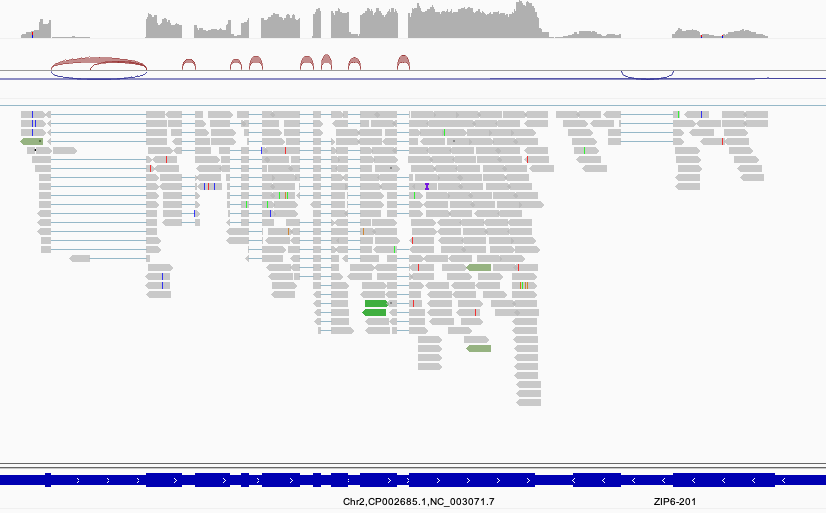
結果をIGV等のゲノムブラウザで可視化すると以下のようにマッピングされた様子が確認できます。
論文に必要な解析が簡単にできるRNA-Seqデータ解析ツール
以下のような方にオススメのRNA-Seqデータ解析ツールです。
✔︎ 他社のソフトウェアを試したが操作が難しいと感じた
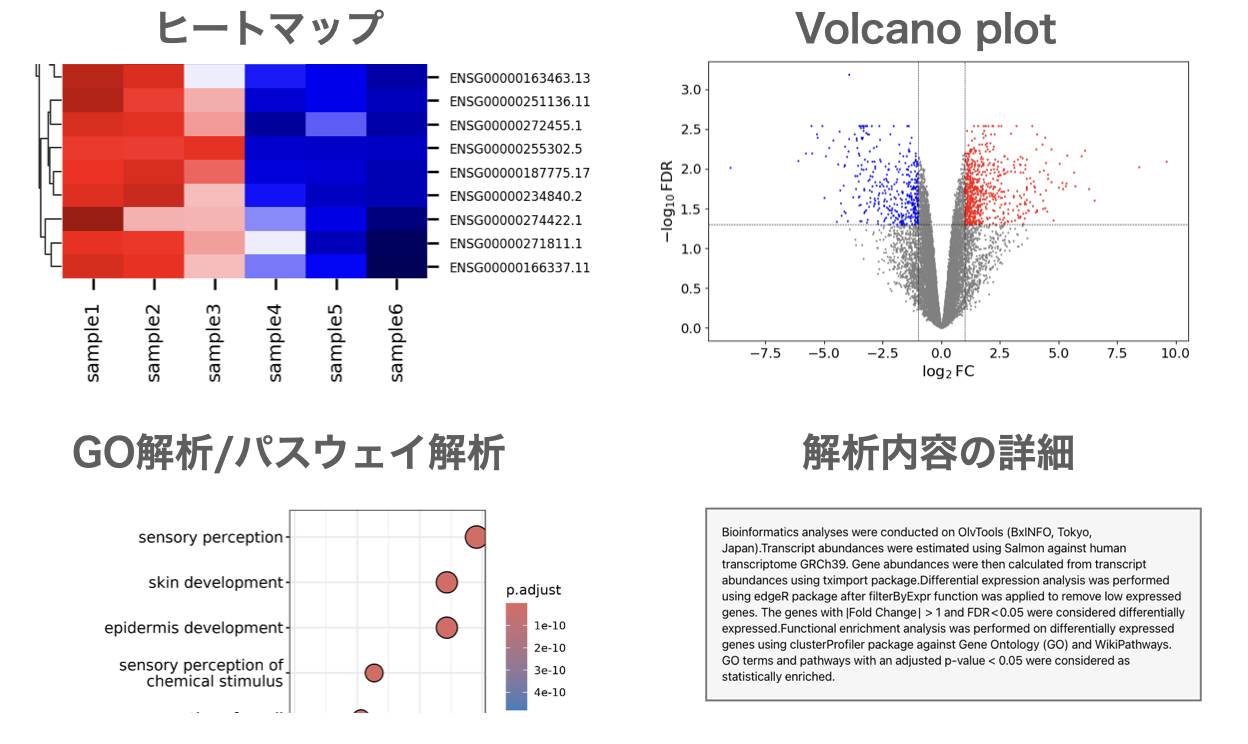
遺伝子発現量の定量、発現変動遺伝子抽出(DEG解析)、Volcano plot描画、MAプロット描画、ヒートマップ描画、GO解析、パスウェイ解析等 を簡単に実施できます。

合同会社BxINFO
バイオインフォマティクスを専門とする研究支援企業です。
RNA-Seq解析を中心に、ライフサイエンスの研究に役立つツール・情報を提供しています。
→ 詳しくはこちら