【FastqQCの使い方】Fastqファイルのクオリティチェック
はじめに
次世代シーケンサー(NGS)を用いたシーケンスを行うと、FASTQファイルと呼ばれるリードの塩基配列とクオリティスコアが書かれた生データが得られます。次世代シーケンス(NGS)を実施したら、まずはFASTQファイルのクオリティチェックを行うことで、リードの品質に問題がないか確認する必要があります。FASTQファイルのクオリティチェックのソフトウェアとして最も有名であるのがFastQCです。
本ページでは、FastQCを用いてたクオリティチェックの手順を解説していきます。
インストール
こちらからインストール可能です。
Macを使用している方でもCUIでの操作を行う場合には、Win/Linux zip fileを選択するのが良さそうです。
以下のようにコマンド操作でダウンロードしても大丈夫です。(バージョンは適宜変更してください。)
うまく動作するか、ヘルプメッセージを表示してみます。
以下のように表示されていれば成功です。fastqcにパスを通しておくと良いでしょう。

実行
resultsフォルダの中にHTMLファイルとZIPファイルが作成されていれば成功です。
結果
HTMLファイルは以下のような内容です。
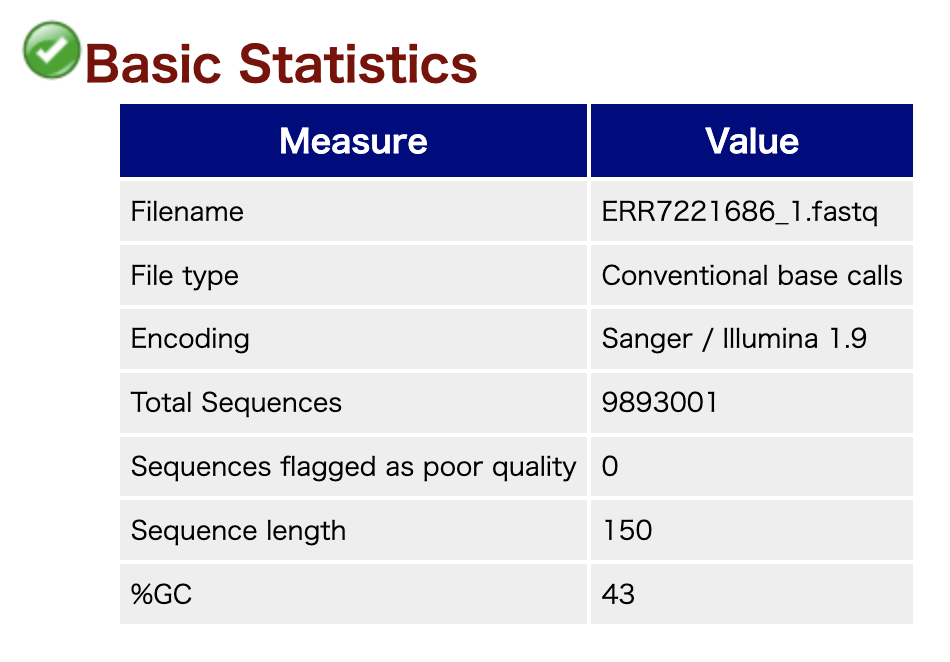
Basic Statistics
基本的な情報が表示されます。
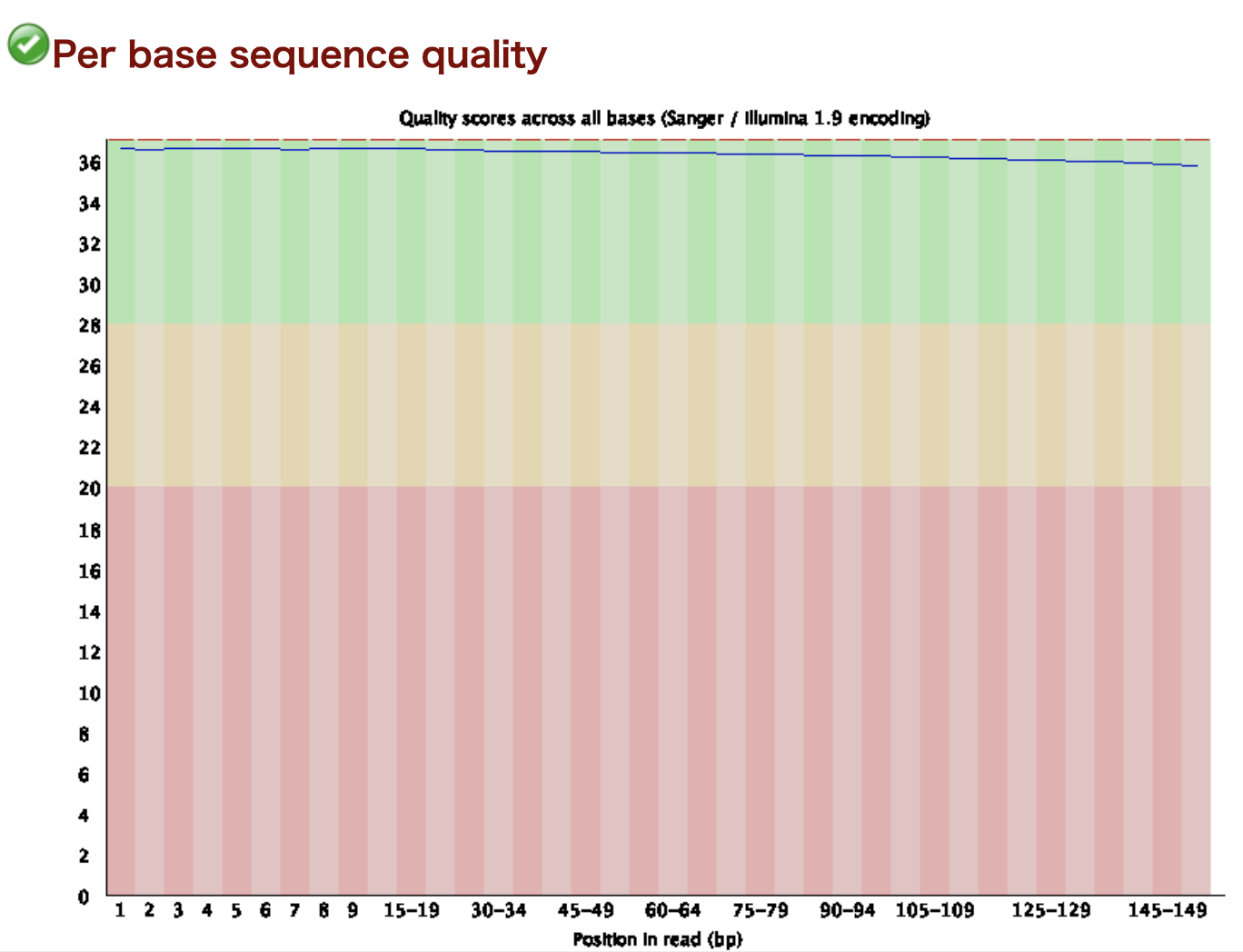
Per base sequence quality
リードのポジションごとのクオリティが表示されます。横軸がリードでのポジションで縦軸がクオリティです。
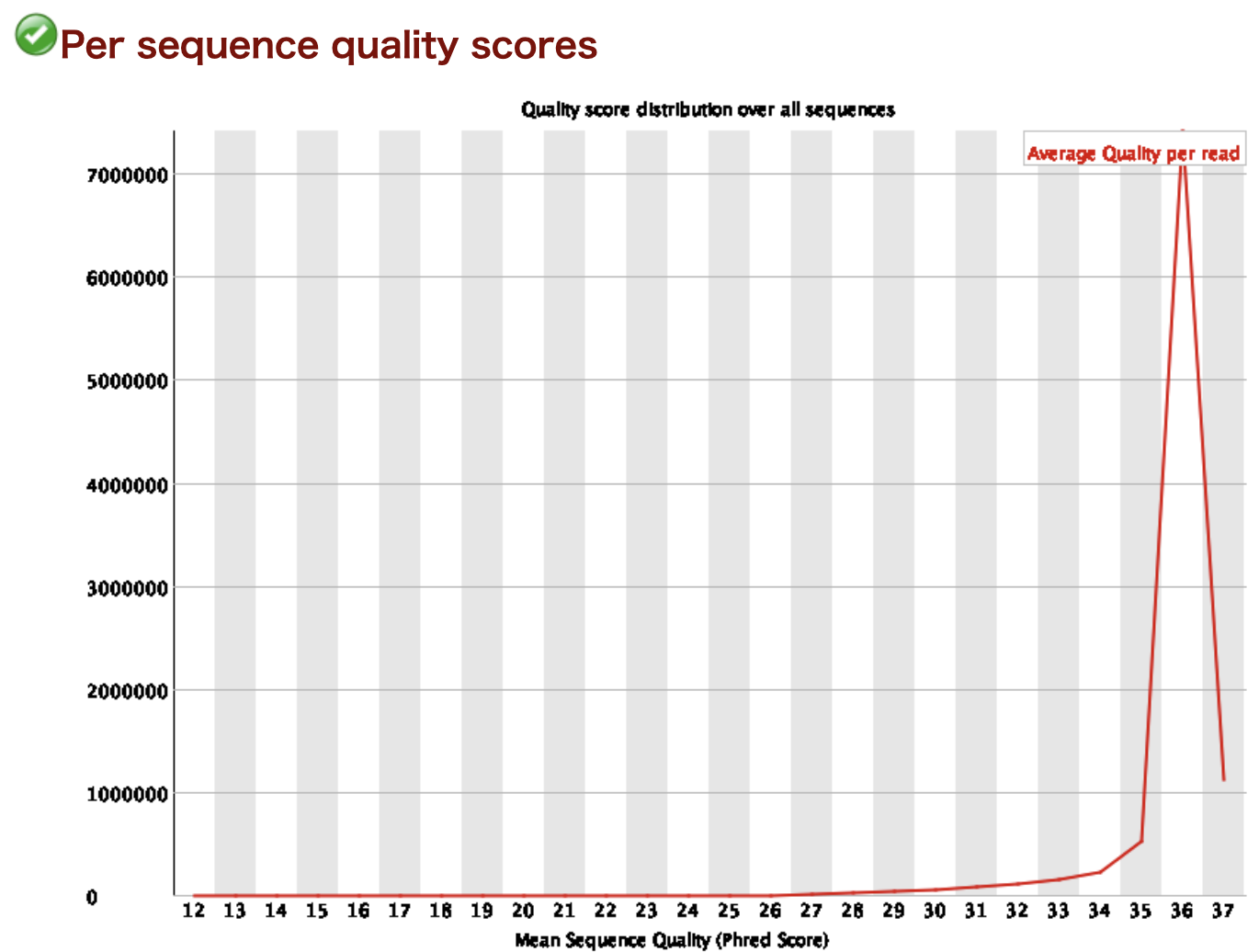
Per sequence quality scores
平均クオリティスコアの分布です。横軸が平均クオリティスコアで縦軸がリードの個数です。
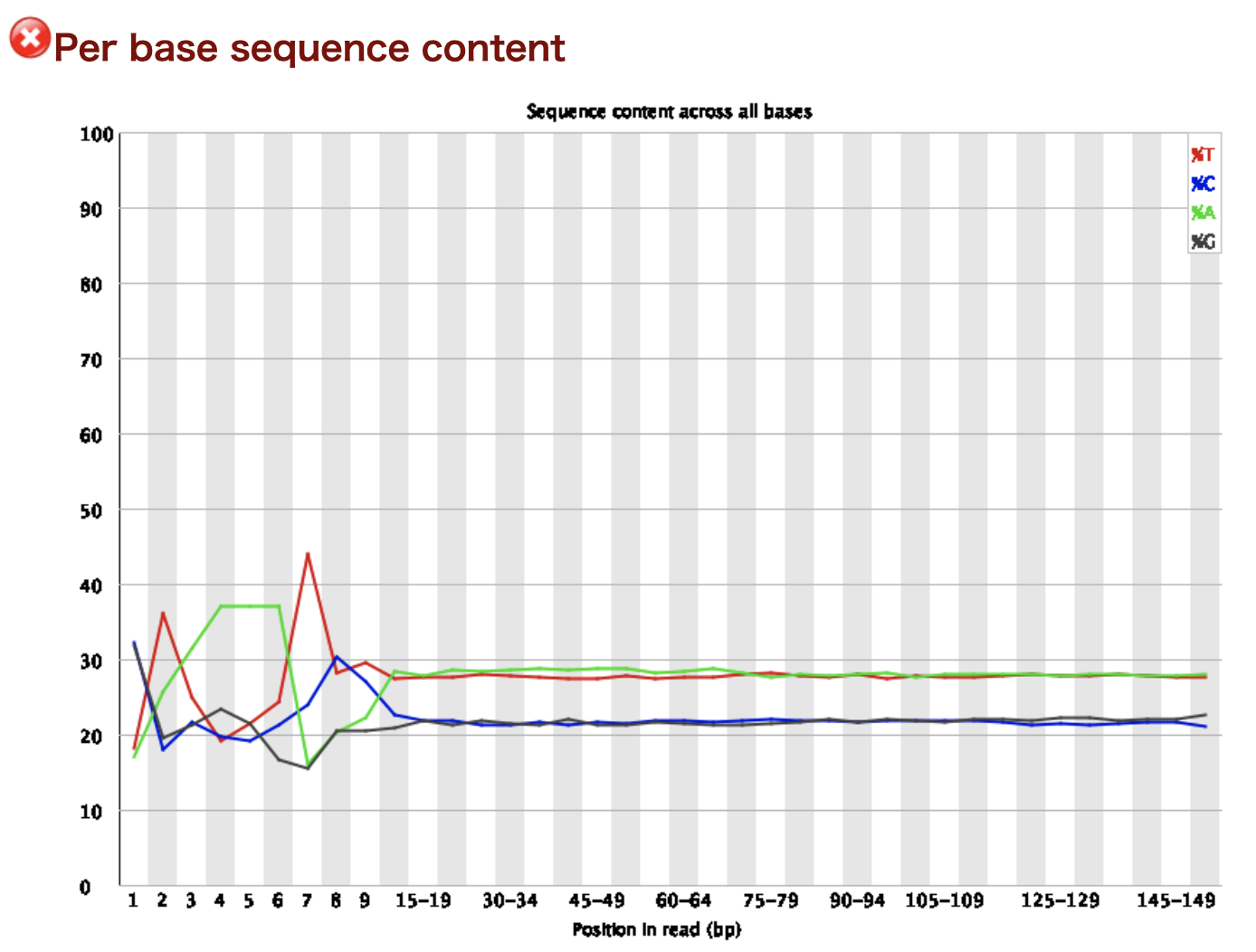
Per base sequence content
リードのポジションごとの塩基の割合です。横軸がリードのポジションで縦軸が割合です。
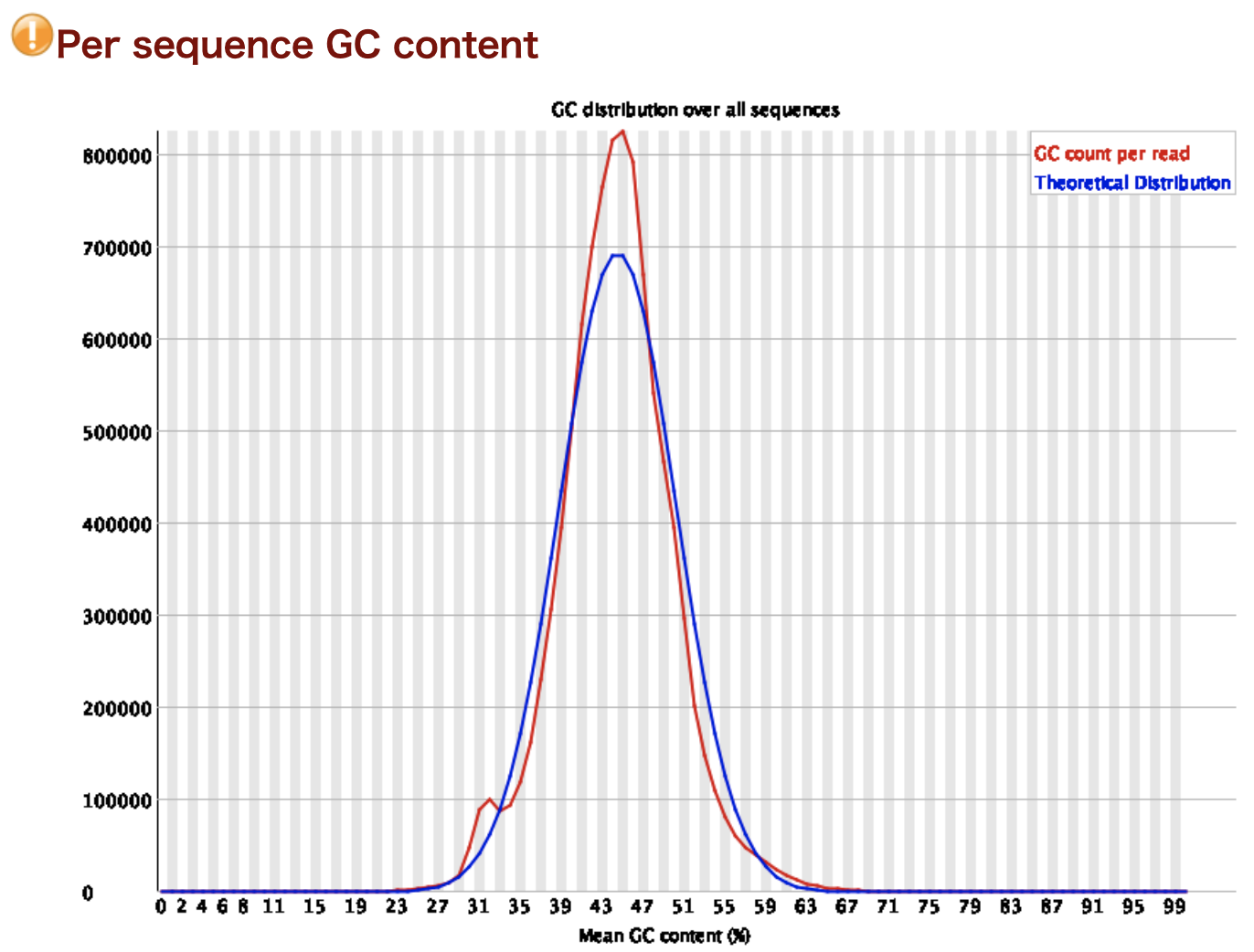
Per sequence GC content
リードごとのGC含量の分布です。横軸がGC含量で縦軸がリードの個数です。
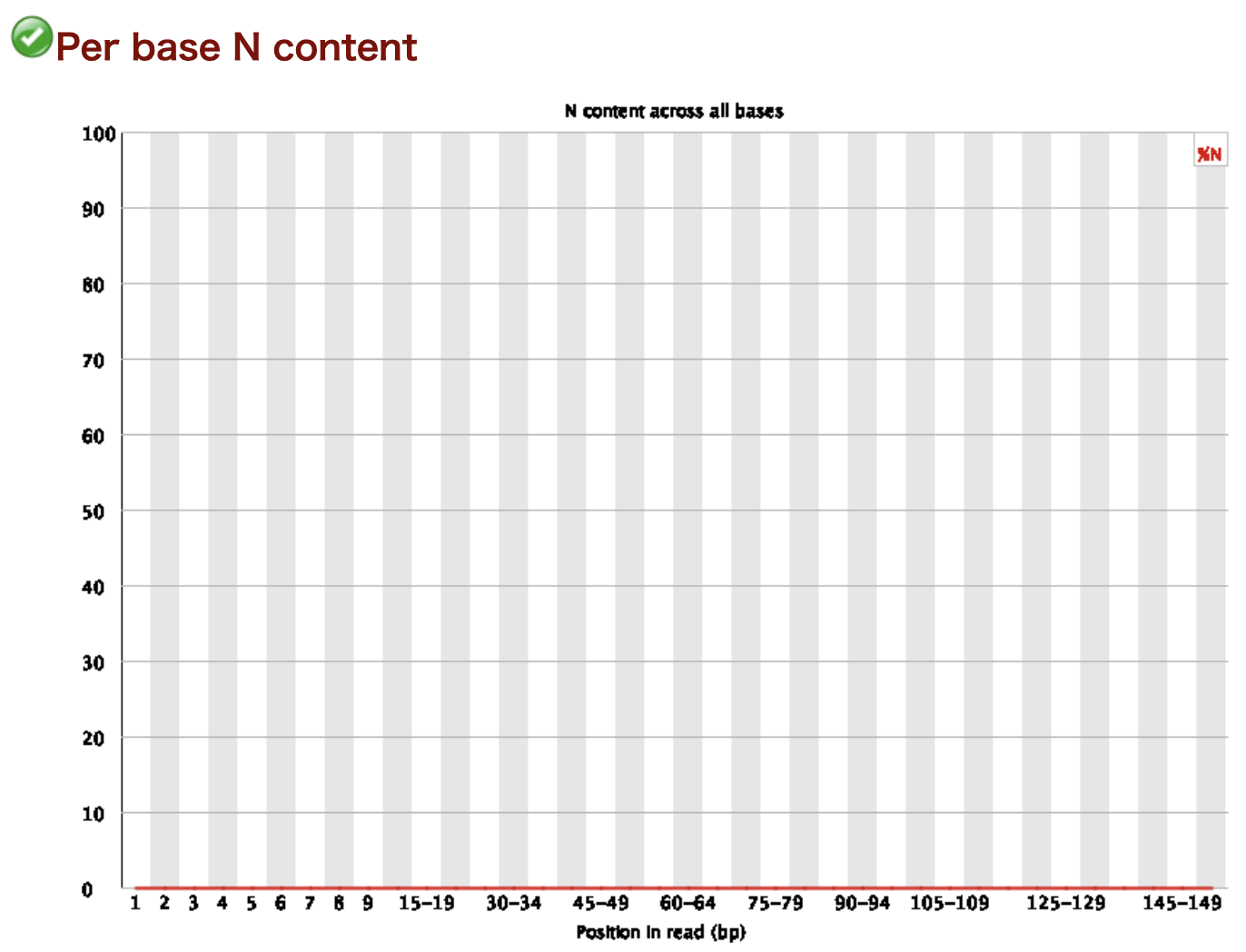
Per base N content
リードのポジションごとのNの割合です。横軸がリードのポジションで縦軸が割合です。
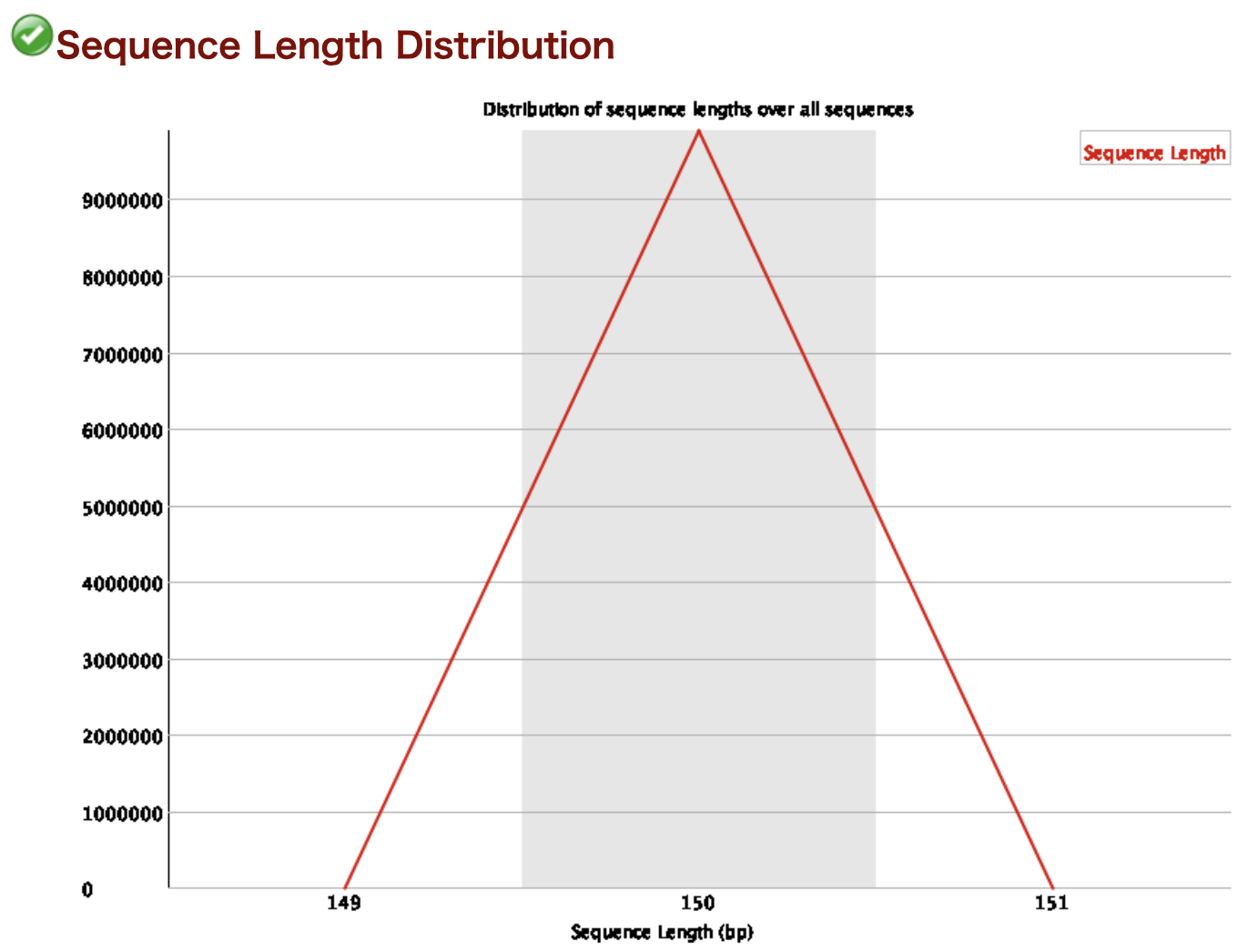
Sequence Length Distribution
リード長の分布です。横軸がリード長で縦軸がリードの個数です。
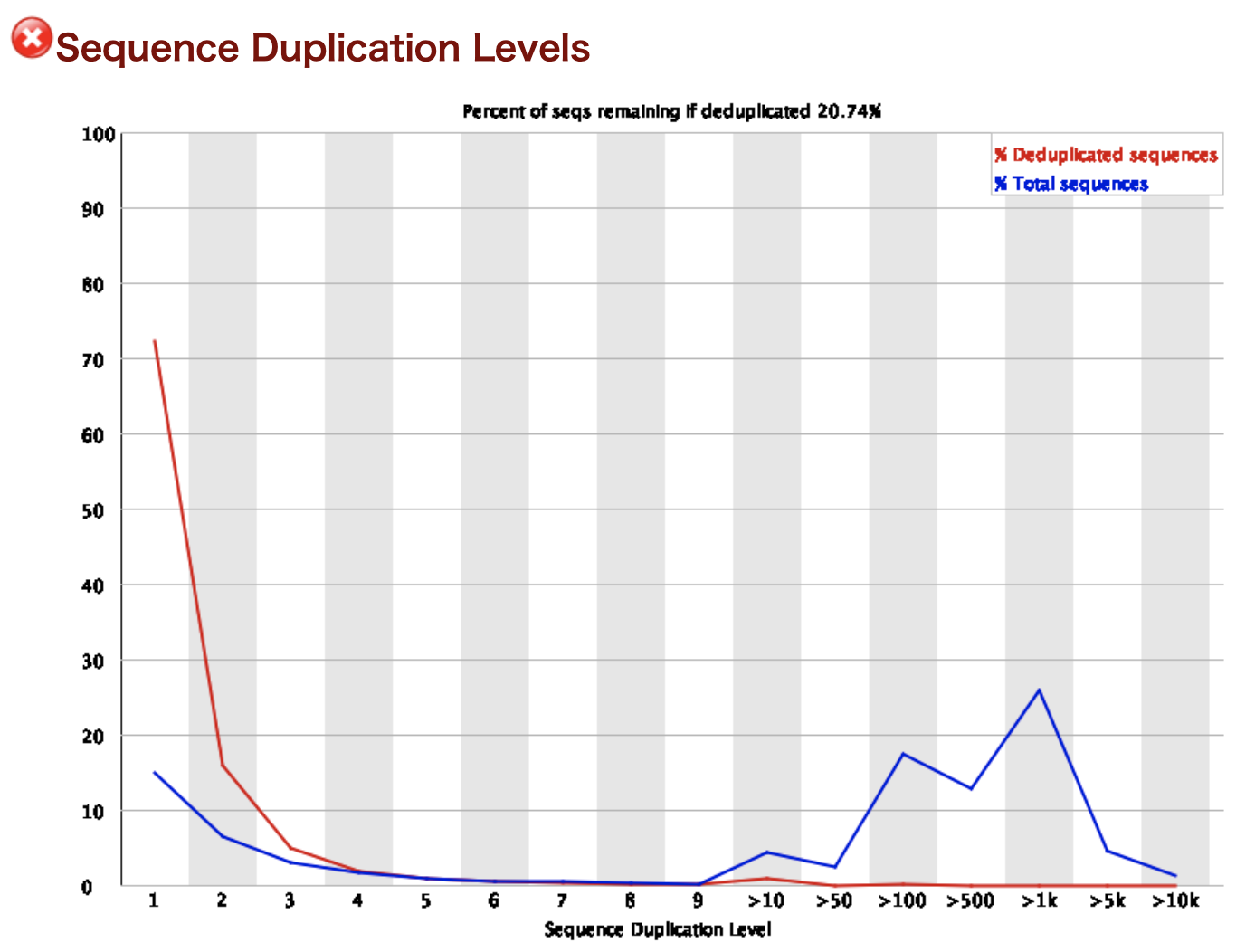
Sequence Duplication Levels
リードの重複の度合いを示しています。横軸が重複の回数で縦軸が重複しているリードの割合です。
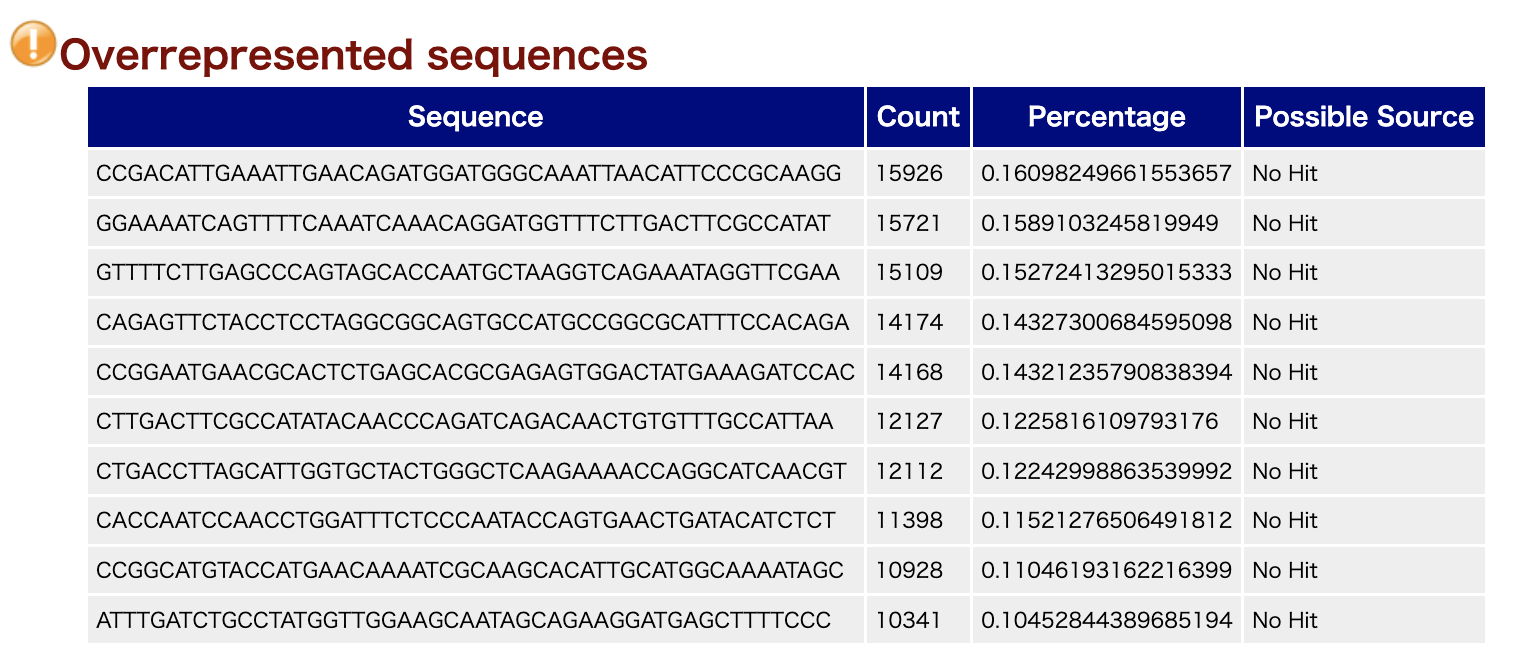
Overrepresented sequences
多く出現する配列が表示されます。
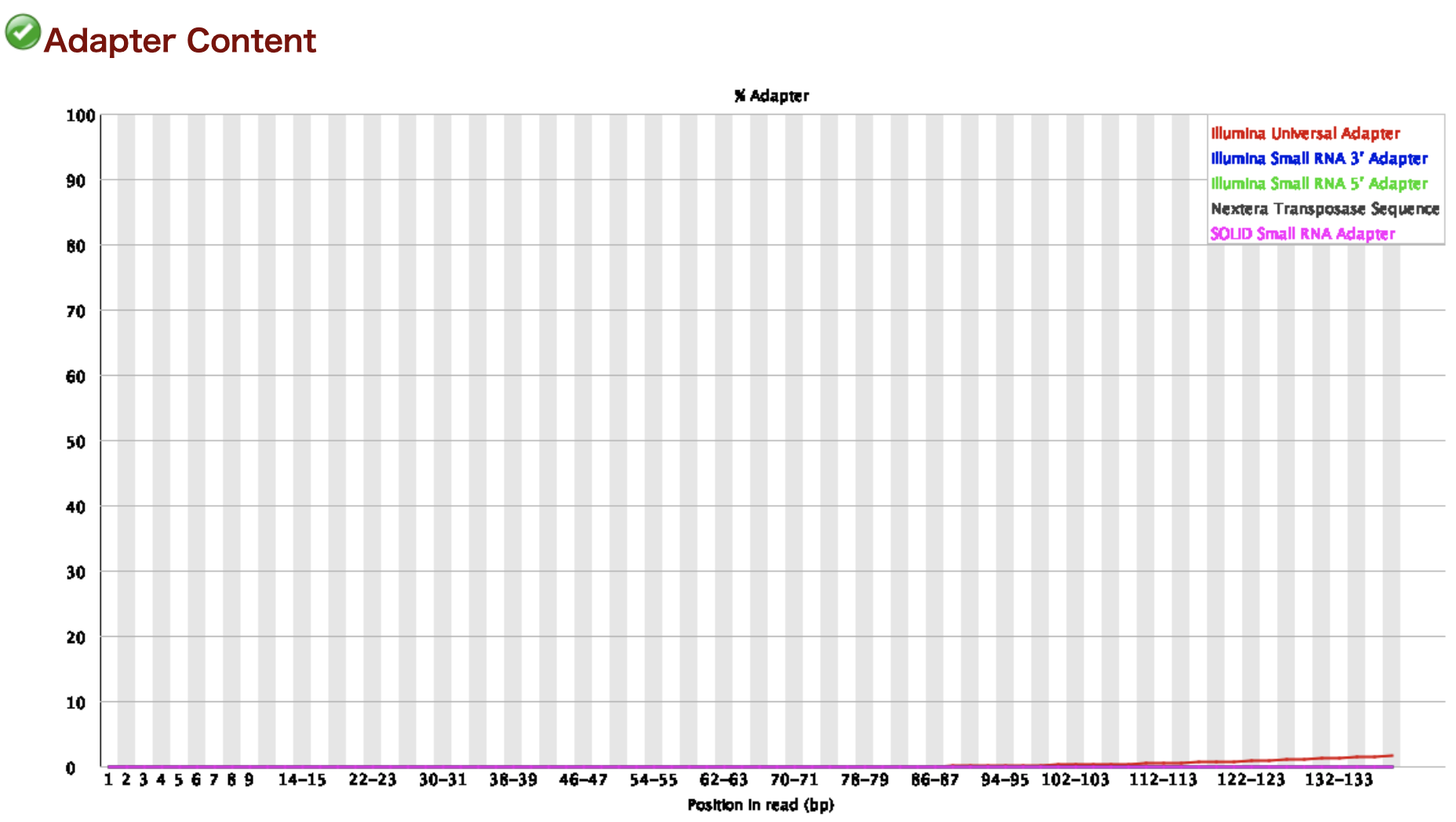
Adapter Content
リードのポジションごとのアダプター配列が存在する割合です。横軸がリードのポジションで縦軸が割合です。
論文に必要な解析が簡単にできるRNA-Seqデータ解析ツール
RNA-Seqデータ解析ツールを利用すれば、外部委託や共同研究者への依頼は必要ありません。高スペックなコンピュータの準備やLinuxコマンドの操作も不要ですので、いますぐにご自身で解析できるようになります。
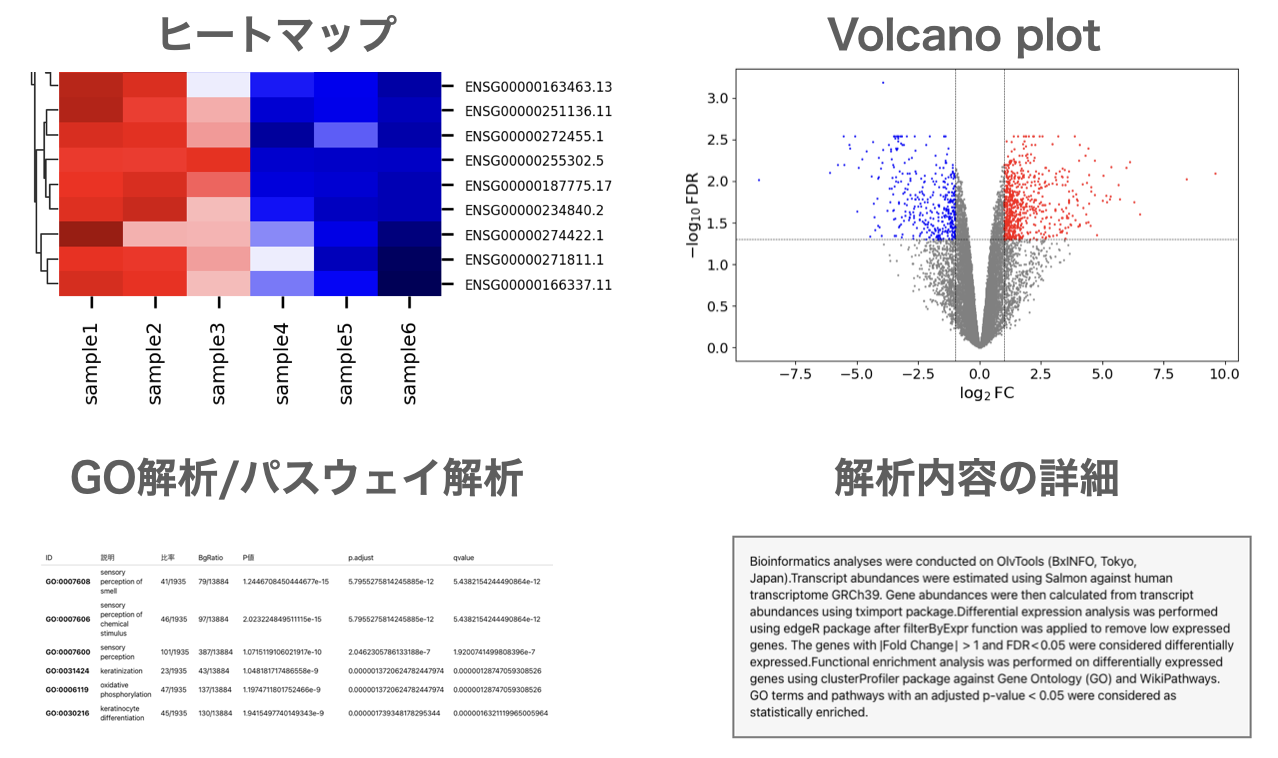
遺伝子発現量の定量、発現変動遺伝子抽出(DEG解析)、Volcano plot描画、MAプロット描画、ヒートマップ描画、GO解析、パスウェイ解析等 を簡単に実施できます。